M.D Neuroscience/Neurogenomics/bioinformatics 整理與分享有趣期刊
Metagenomics (總體基因體學) 介紹
什麼是Metagenomics?
總體基因組學是將現代基因組技術應用於直接在自然環境中研究微生物群落,無需對單個物種進行隔離和實驗室培養。
在土壤、海洋、泥土、森林、太空甚至人體中都可以找到不同的微生物群落。在這些環境中可以發現各種微生物(細菌、病毒/噬菌體、真核生物蠕蟲和線蟲)。
Why Metagenomics?
研究微生物的老方法是拿樣本放在培養皿中,看看是否有任何生長,分離培養物。然而,多數細菌種類不能用這種傳統方式培養。因此,在總體基因組學中,我們所做的是直接從微生物群落中獲取樣本並對其進行測序。
Metagenomics中的兩個主要問題
每個宏基因組學研究都需要釐清兩個主要問題
- Who is there?
- What are they doing?
我們想確定樣本中存在哪些微生物種類,以及確定他們的功能
兩種常見用於測序的方法
- Targeted metagenomics: Targeted基因組的特定區域(ex:16S rRNA 和 18S rRNA),這些區域在多個生命體和sample中都有存在。這種方法提供了更精確的數據和更多的深度,但可能會導致某些目標區域的放大不均。
- Shotgun metagenomics: 可以對樣本中的所有內容進行測序。適用於所有生物。它為遺傳物質(尤其是DNA )提供了更高的分辨率,但會產生較複雜的dataset。
Shotgun metagenomics
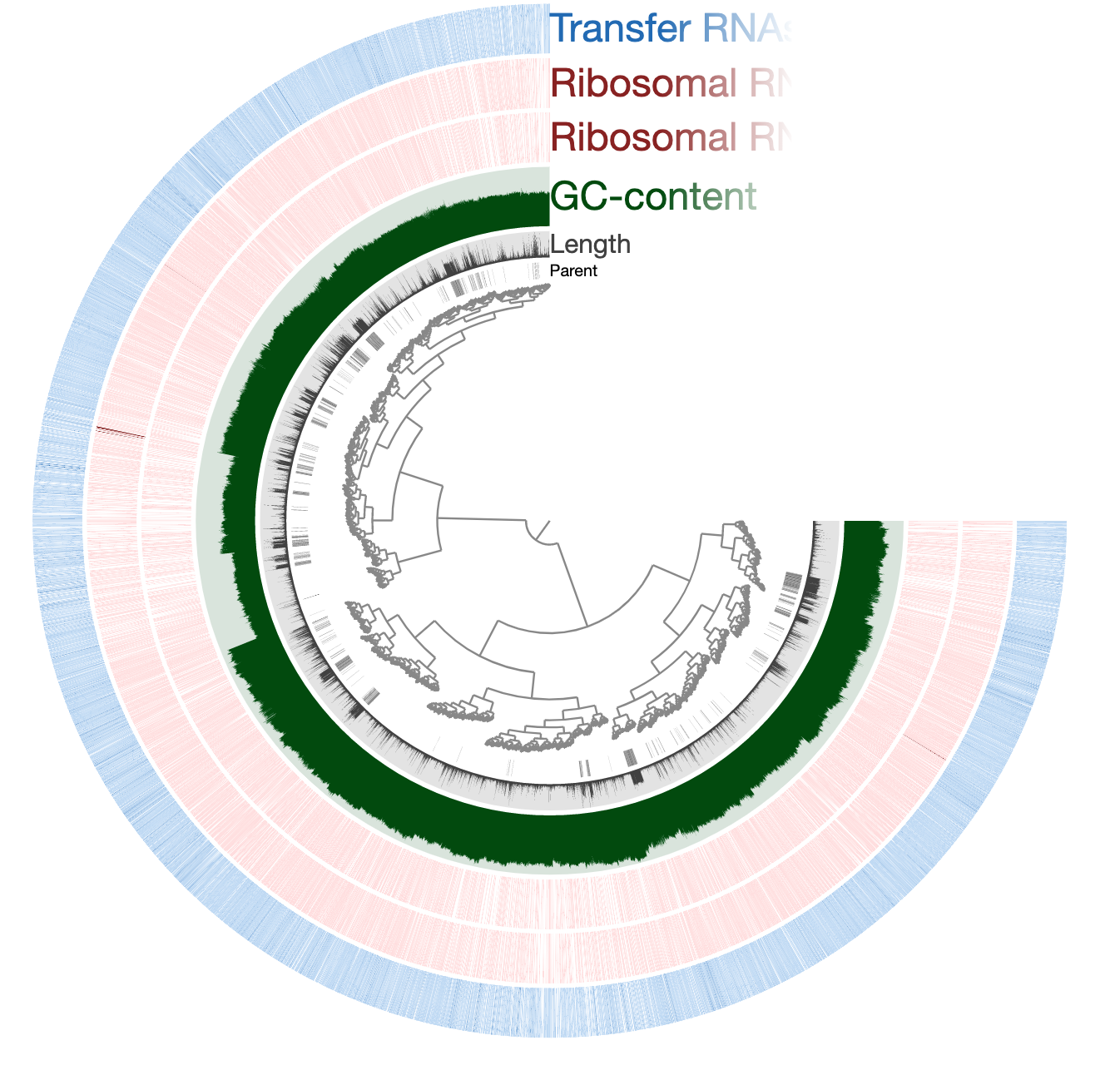
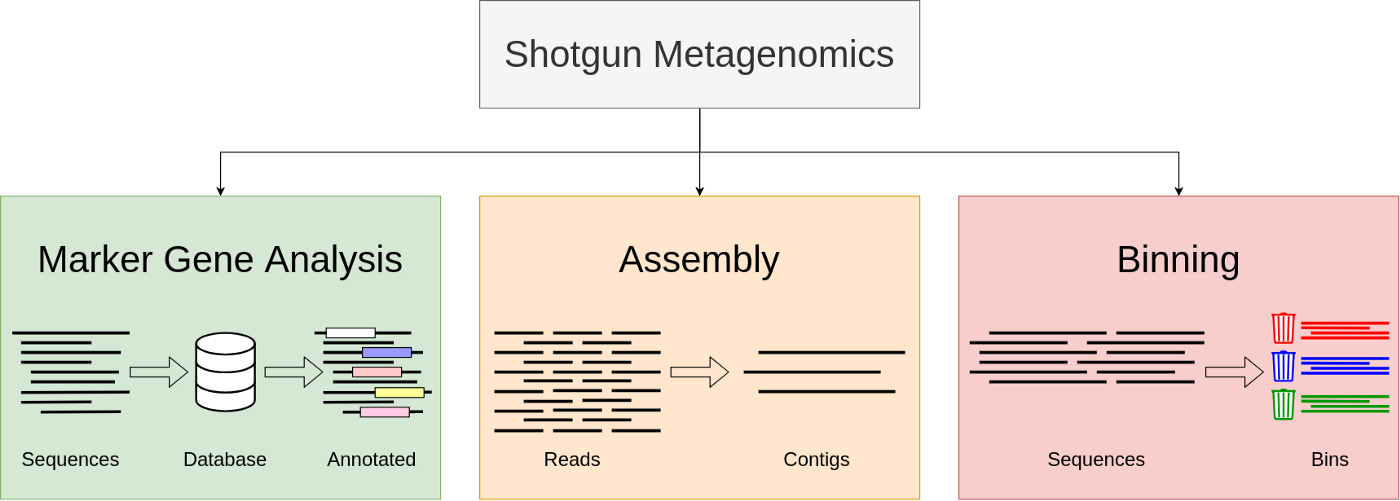
處理metagenomics數據集時有三種主要方法 ,如下圖所示
- Marker gene analysis:將序列與Marker gene數據庫進行比較,比較它們的相似性並對序列進行分類註釋。最常用的標記基因是ribosomalRNA基因,它們具有single copy的特性並且在微生物基因組中很常見。
- Binning:將序列聚類成與分類群(taxonomic groups, 如物種、屬)相對應的群。
- Assembly:將樣本中的所有小序列放在一起,形成更長序列。
Targeted Metagenomics
從樣本中提取遺傳物質,並根據感興趣的區域對感興趣的基因進行 PCR 。最常用的基因是16S RNA。該基因被稱為“universal phylogenetic marker”。它存在於所有微生物中,並且包含一個單拷貝基因(single copy)。

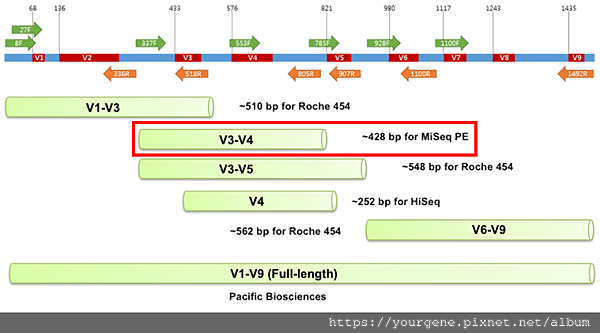
16S rRNA位在原核細胞的核醣體小次單元(Ribosomal Small Subunit; SSU)上
其序列包含9個高變區(V1~V9)及10個保守區(Conserved Regions),如下圖圖所示。利用16S rRNA的這些高變區去做定序,可以鑑定微生物的種類。
ref:
https://hackmd.io/@whYPD8MBSHWRZV6y-ymFwQ/SyHmXjD_P
http://www.yourgene.com.tw/content/messagess/contents/655412454415503242/
http://www.yourgene.com.tw/content/messagess/contents/655643254701452640/
喜欢我的文章吗?
别忘了给点支持与赞赏,让我知道创作的路上有你陪伴。
发布评论…